New In 
Lasso pour les modèles à risques proportionnels de Cox
Si vous disposez de données temporelles, également connues sous le nom de données de temps de survie ou de temps de défaillance, et de nombreux prédicteurs, consultez les outils lasso, cox et elasticnet.
(Et lorsque nous parlons de nombreux prédicteurs, nous voulons dire des centaines, des milliers ou plus ! (Et lorsque nous parlons de nombreux prédicteurs, nous voulons dire des centaines, des milliers ou plus !) Nouvellement dans Stata 18, ces commandes étendent la suite lasso existante pour la prédiction et la sélection de modèles afin d’inclure un modèle de risques proportionnels de Cox semi-paramétrique à haute dimension.
Après lasso cox et elasticnet cox, vous pouvez utiliser stcurve pour tracer la fonction de survie, d’échec, de hasard ou de hasard cumulatif ou utiliser l’un des autres outils de post-estimation disponibles après lasso et elasticnet.
Let’s see it work
Nous illustrons lasso cox avec un exemple qui prédit le risque de décès pour les patients atteints d’un adénocarcinome pulmonaire de stade I. L’adénocarcinome pulmonaire est l’un des cancers du poumon non à petites cellules les plus courants.
L’adénocarcinome de stade I indique que la taille de la tumeur est relativement petite et que le cancer ne s’est pas propagé à d’autres organes distants. Les patients atteints d’adénocarcinome de stade I ont des résultats de survie variés, même s’ils sont au stade précoce du développement du cancer. Par exemple, Yu et al. (2016) montrent que, dans une cohorte, plus de 50 % des patients atteints d’adénocarcinome de stade I sont décédés dans les 5 ans suivant le diagnostic initial, tandis qu’environ 15 % des patients ont survécu plus de 10 ans.
Les caractéristiques des images histopathologiques sont utilisées pour l’analyse pronostique. Nous pouvons utiliser lasso cox pour extraire les principales caractéristiques des images histopathologiques qui permettent de distinguer les survivants à court terme des survivants à long terme.
Nous disposons d’un ensemble de données fictives sur la survie (lungcancer.dta) inspiré par Yu et al. (2016). La variable t enregistre soit le moment du décès, soit la censure en mois pour les patients atteints de cancer du poumon adénocarcinome de stade I. La variable indicatrice died est 1 ou 0 si le patient est décédé ou censuré, respectivement. Il y a 500 caractéristiques d’images histopathologiques, histfeature1 à hisfeature500, et seulement 250 patients. L’analyse vise à classer un nouveau patient dans un groupe à faible risque ou à risque élevé, en fonction des caractéristiques de l’image histopathologique.
Nous chargeons d’abord l’ensemble de données, puis nous tapons stset pour montrer qu’il a déjà été stset.

Ensuite, nous divisons l’échantillon complet en données de formation et de test. Les données d’entraînement seront utilisées pour l’estimation, et les données de test seront utilisées pour mesurer la performance de la prédiction.
Nous utilisons splitsample pour diviser les données en deux parties. Nous utilisons l’option generate(group) pour créer une nouvelle variable, group, qui est égale à 1 si elle appartient aux données d’entraînement ou à 0 si elle appartient aux données de test. L’option split(0.6 0.4) spécifie que 60 % des données entières sont utilisées comme données d’entraînement et 40 % comme données de test. Pour que les résultats soient reproductibles, nous spécifions l’option rseed().
. splitsample, generate(group) split(0.6 0.4) rseed(12345)
Pour une utilisation ultérieure, nous enregistrons les données d’entraînement sous le nom de lungcancer_training.dta et les données de test sous le nom de lungcancer_testing.dta.
. preserve . keep if group == 1 (100 observations deleted) . save lungcancer_training, replace file lungcancer_training.dta not found file lungcancer_training.dta saved . restore . preserve . keep if group == 2 (150 observations deleted) . save lungcancer_testing, replace file lungcancer_testing.dta not found file lungcancer_testing.dta saved . restore
Nous allons maintenant ajuster un modèle lasso cox en utilisant uniquement les données d’apprentissage. Par défaut, nous utilisons la validation croisée. Nous spécifions rseed() pour la reproductibilité.
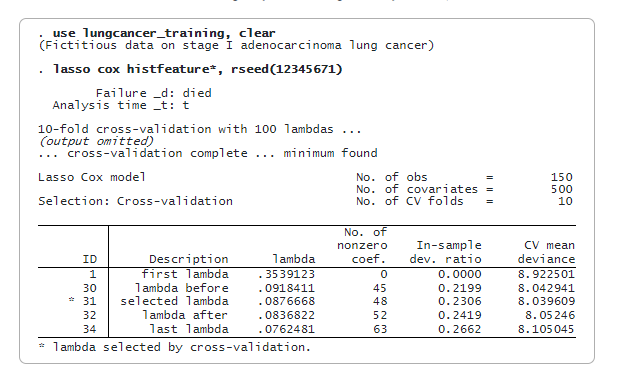
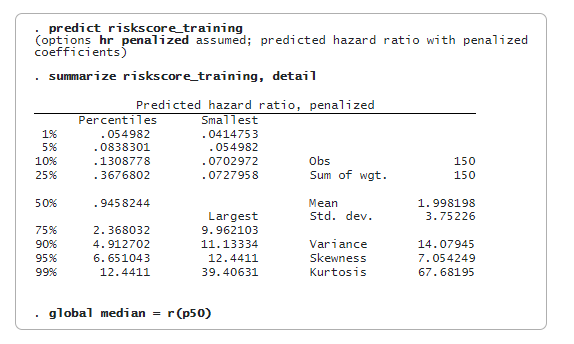
lasso cox sélectionne 48 des 500 caractéristiques. Nous pouvons maintenant prédire le rapport de risque relatif pénalisé (variable riskscore_training) et évaluer les scores de risque. Nous utiliserons la médiane de riskscore_training comme seuil pour classer un patient comme étant à faible risque ou à risque élevé. Nous stockons la valeur médiane dans une macro globale (median) pour une utilisation ultérieure.
Nous utilisons maintenant les données de test pour valider le modèle. Tout d’abord, nous prédisons le ratio de risque pénalisé (variable riskscore_testing) dans l’échantillon de test. Ensuite, nous comparons riskscore_testing à la médiane du rapport de risque obtenu dans les données d’apprentissage ($median). Le patient est étiqueté comme présentant un risque élevé si le score de risque prédit est supérieur ou égal à la médiane. Le patient est classé à faible risque si le score de risque prédit est inférieur à la médiane.
. use lungcancer_testing, clear (Fictitious data on stage I adenocarcinoma lung cancer) . predict riskscore_testing (options hr penalized assumed; predicted hazard ratio with penalized coefficients) . generate byte risk = (riskscore_testing >= $median) . label define risk_lb 1 "High risk" 0 "Low risk" . label values risk risk_lb
Pour évaluer l’efficacité de la classification des risques, nous examinons tout d’abord le diagramme de Kaplan-Meier, qui trace la courbe de survie pour les groupes à faible risque et à risque élevé.
. sts graph, by(risk)

Le graphique montre que les patients prédits à haut risque ont une courbe de survie plus abrupte que les patients prédits à faible risque. Pour confirmer cette conjecture, nous effectuons un test de log-rank.

Le test du log-rank rejette l’hypothèse selon laquelle les patients prédits à faible risque et à haut risque ont les mêmes fonctions de survie. Le diagramme de Kaplan-Meier et le test du log-rank montrent que l’utilisation de la médiane des rapports de risque prédits permet de distinguer efficacement un patient à faible risque d’un patient à haut risque. Nous pouvons maintenant faire des prédictions pronostiques à partir de nouvelles données.
L’ensemble de données (newlungcancer.dta) contient des caractéristiques d’images histopathologiques pour quelques nouveaux patients atteints d’adénocarcinome de stade I, et nous n’avons pas encore observé leur temps de survie parce qu’ils sont encore en vie. Sur la base du modèle de prédiction de lasso cox, nous voulons classer ces nouveaux patients comme étant à faible risque ou à risque élevé. Pour atteindre cet objectif, nous devons prédire uniquement les rapports de risque des nouveaux patients et les comparer au niveau médian du score de risque obtenu dans les données d’apprentissage.

Le tableau du niveau de risque prédit montre que 27 patients sont classés comme présentant un risque faible, tandis que 23 patients sont classés comme présentant un risque élevé.
Référence
Yu, K., C. Zhang, G. J. Berry, R. B. Altman, C. Ré, D. L. Rubin, et M. Snyder. 2016. Predicting non-small cell lung cancer prognosis by fully automated microscopic pathology image features (Prédire le pronostic du cancer du poumon non à petites cellules par des caractéristiques d’image de pathologie microscopique entièrement automatisées). Nature Communications 7(12474).
